动植物基因组中包含大量的调控元件,确定调控元件在基因组上的位置和解析其功能一直是生命科学领域的研究热点。2003年,全球科学家联手发起ENCODE计划,旨在绘制人类及小鼠基因组功能元件的综合图谱。在第二阶段的ENCOD计划中引入了基于高通量测序的组学技术,如ChIP-seq、DNase-seq等。随着高通量测序技术的迅猛发展,调控组学数据呈爆炸式增长。如今,ENCODE官网已经更新至第五版,为研究人员提供宝贵的数据资源。然而,植物研究领域还没有类似于ENCODE计划的数据资源库,极大限制了植物功能基因组学的研究和应用。越来越多的研究表明,植物基因组调控区富集大量的农艺性状和作物驯化关联位点,靶向编辑调控元件可以精准改变重要农艺性状基因的表达变化,因此,建立植物调控基因组参考图谱将为作物遗传精准改良提供重要的资源。
为了给广大植物研究学者提供一个系统全面的调控基因组资源数据库及分析平台,生命科学学院陈迪俊课题组收集了超过40个物种,总量大于10,000套植物调控组学数据,其中包括ChIP-seq、DAP-seq、DNase-seq和ATAC-seq等。我们采用ENCODE计划推荐的数据分析标准,对来自世界各地不同实验室的数据进行人工注释并统一分析处理,系统评估数据质量,并将数据资源和分析结果整合到ChIP-Hub(https://biobigdata.nju.edu.cn/ChIPHub/ )在线数据库中。该研究不仅能够帮助植物科研学者快速定位植物基因组上潜在的调控元件,并提供了便利的信息检索和数据分析可视化平台,为作物精准改良提供了宝贵的参考资源。
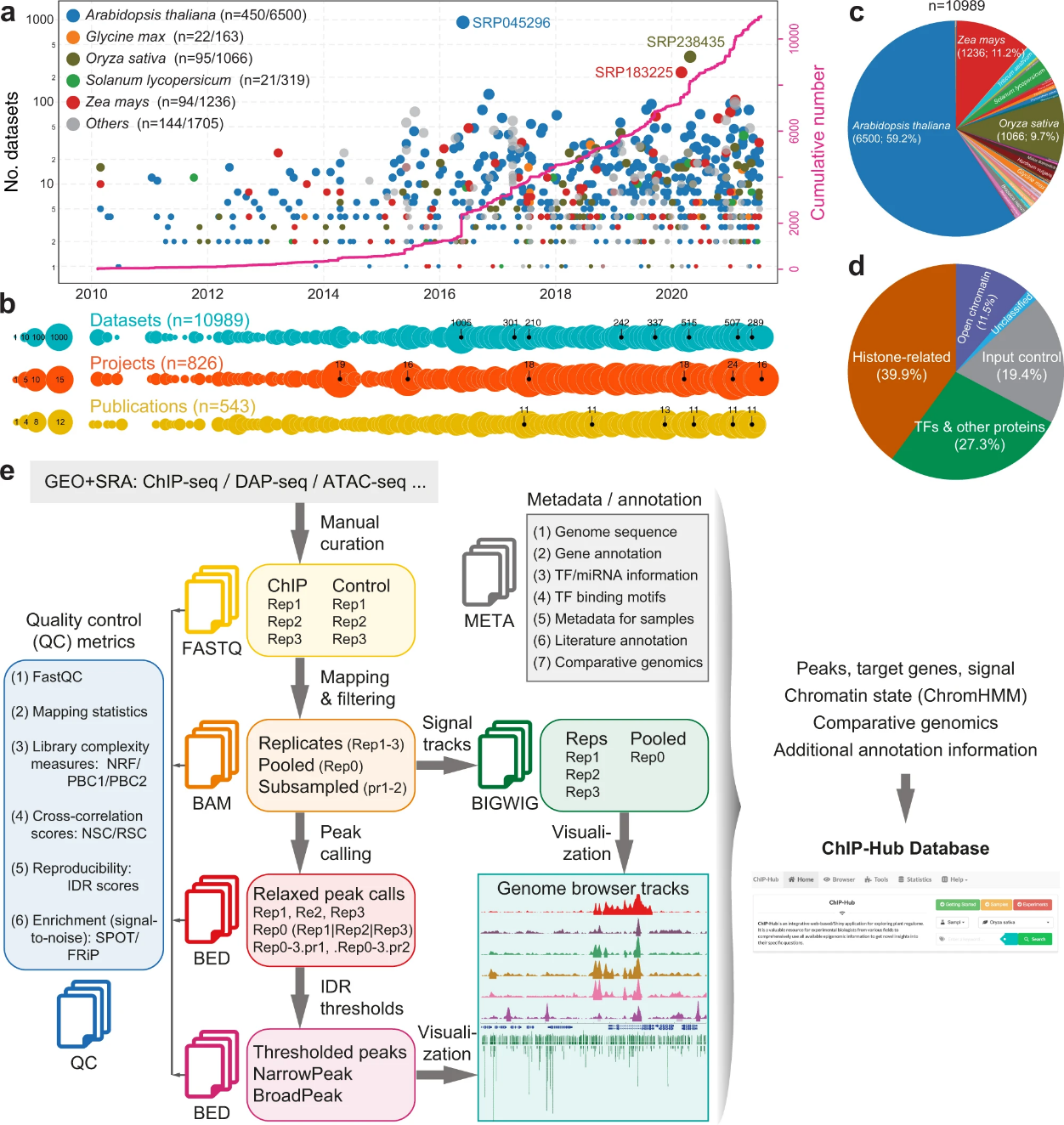
图1.a-d 所收集的植物调控组学数据概览;e 调控组学数据分析流程示意图
此外,研究者还将收集到的数据进行整合分析。首先使用模式植物拟南芥的转录因子(TF)ChIP-seq数据分析后发现,尽管这些数据产生于不同的实验条件,但是仍能观察到不同TF之间存在协同作用模块进而共同调控下游靶基因。在此基础上,进一步构建并分析了miRNA和转录因子协调作用的大尺度基因调控网络和网络基序,通过文献调研对预测的调控关系辅以验证。
接着使用拟南芥10种不同组织的染色质可及性数据(ATAC-seq或DNase-seq),鉴定到了一系列的组织特异性的启动子和增强子,总体上讲,增强子的组织特异性要高于启动子。与此相对应,基于比较调控基因组学的分析发现增强子序列的进化速率比启动子要快。
最后通过整合不同层次的调控组学数据,发现由多种组蛋白修饰数据(HM ChIP-seq)定义的染色质状态在染色质开放性(ATAC-seq或DNase-seq)、转录因子结合位点富集(TF ChIP-seq)、基因表达(RNA-seq)以及序列保守性等方面存在很强的相关性;同时,通过比较拟南芥、水稻、玉米、大麦和小麦等不同基因组的染色质状态,发现具有较高转录活性的染色质状态在不同物种之间更加保守。以上分析表明,植物基因组中可能存在一套由调控序列决定的精准控制基因表达时空特异性的"调控密码子"。
该研究成果于2022年6月14日以题为ChIP-Hub provides an integrative platform for exploring plant regulome的研究论文在Nature Communications在线发表,并且被杂志选为Editors’ Highlights文章。生命科学学院陈迪俊副教授为最后通讯作者,德国柏林洪堡大学Kerstin Kaufmann教授为共同通讯作者。生命科学学院傅靓彧,博士研究生祝涛,硕士研究生周欣恺和于冉冉为该论文的共同第一作者。浙江大学生命科学学院陈铭教授和张霈婧博士对本研究亦有贡献。本研究得到了国家自然科学基金和37000cm威尼斯登峰人才支持计划的资助。作者特别感谢37000cm威尼斯高性能计算中心为本研究提供了宝贵的高性能计算(HPC)资源,37000cm威尼斯信息中心为本研究提供了技术支持。

论文链接:https://doi.org/10.1038/s41467-022-30770-1