植物与病原“军备竞赛”式的相互作用是驱动植物适应性演化的重要动力之一。在数亿年的演化过程中,植物应对环境中复杂多变的病原环境而形成了一类具有特殊功能的基因——植物抗病基因,NLR(核苷酸结合位点富含亮氨酸重复序列受体)基因是其中数量最多、功能最重要的一个基因家族,包括RNL、TNL和CNL三个不同的功能亚类。然而,随着植物的起源和发展,在适应不同病原环境时,植物NLR基因是如何演化的? 其不同的功能亚类之间存在怎样的演化关系?这些重要问题一直都不清楚。
近日,37000cm威尼斯生命科学学院陈建群/邵珠卿团队通过对被子植物基因组的大尺度演化分析,揭示了植物抗病基因适应性演化新机制。通过构建迄今为止覆盖被子植物最广泛、包含NLR抗病基因最多的数据库,深入分析NLR基因的演化规律,发现了不同演化分支的被子植物在适应水生、寄生和食虫等特殊生活环境过程中NLR抗病基因显著减少的现象;揭示了信号通路基因的丢失不仅是NLR基因TNL亚类在物种演化中频繁丢失的驱动因素,也是导致NLR基因数量减少的重要内在机制;基于TNL基因与抗病信号传导基因之间频繁的“共丢失”演化模式,发现了多个可能参与NLR抗病基因信号传导的新基因,从演化的角度指导了抗病信号传导新基因的发现。
为了从植物宏观演化尺度上全面分析不同物种中NLR基因及其不同功能亚类的差异,研究者首先以陆地植物最大的演化类群——被子植物为对象,建立了覆盖被子植物37个目85个科305个物种基因组、包含90000多条NLR基因的被子植物NLR基因数据库ANNA(图1, 数据库网址https://biobigdata.nju.edu.cn/ANNA/)。不论是包含物种和基因的数量,还是对被子植物类群的覆盖度,ANNA都是目前为止最为全面的一个植物抗病基因数据库。该数据库为研究人员从NLR基因的结构、功能、演化等角度研究和利用植物抗病基因提供了一个公共服务平台和数据资源。
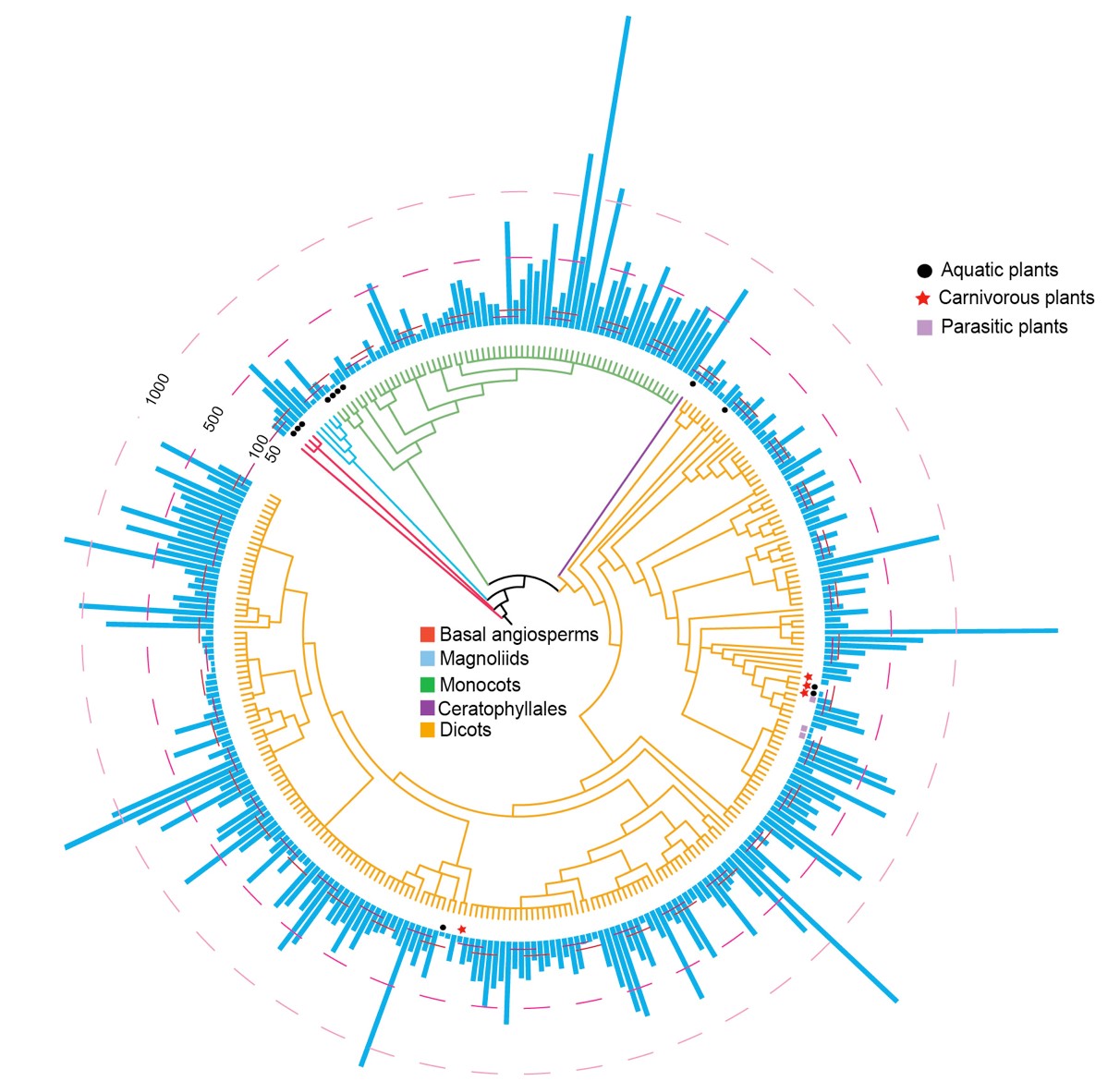
图1. NLR基因在被子植物305个物种中的分布情况。
由于物种演化过程中病原环境的变化往往不可追溯,因此很难确定病原环境变化对物种特异性NLR基因数目变异的影响。该研究借助于ANNA数据库,对不同被子植物的NLR基因数量变异模式进行深入分析发现,在被子植物不同演化分支上,具有水生、寄生和捕虫等特殊生活习性的物种中NLR基因数目显著减少(图1),揭示了植物生活习性变化相关联的NLR基因数量变异。将分析的对象进一步拓展到整个绿色植物(图2A),可以发现:尽管NLR基因的起源可以追溯到绿色植物共同祖先,但是NLR基因的大量扩增却发生在植物登陆之后,这表明NLR抗病基因的扩增确实是植物适应陆地环境中更加复杂的病原选择压力的演化结果;而当陆地植物不同演化分支中的一些物种(如该研究中分析的一些蕨类和多个被子植物物种)重新回到水生环境中,或演化为寄生和食虫的生活习性后,环境中的病原减少或植物根、叶等器官的退化都会使得植物与病原的物理接触减少,进而得以躲避病原的选择压力,导致NLR抗病基因的数量减少。综上,该研究提出了“抗病基因的生态适应演化假说”,即生态适应导致的植物与病原物理接触的减少,降低了对植物免疫系统的选择压力,从而促进了NLR抗病基因的数量减少。
在此基础上,该研究深入探究NLR基因数量变异的驱动力,发现NLR基因中行使病原识别功能的TNL亚类与参与抗病信号传导的RNL亚类的一个演化分支NRG1之间存在密切的协同演化关系,提出NRG1抗病信号通路坍塌驱动了TNL基因在被子植物演化中的多次独立丢失,解析了被子植物TNL基因演化、丢失的分子机制(图2B和C)。此外,研究还发现在大量的水生、寄生和食虫植物中RNL和EDS1家族抗病信号传导基因发生缺失可能导致了这些物种基因组中的NLR基因的数量减少(图2B和C)。上述聚焦NLR抗病信号转导网络的研究,基于演化生物学的基因组大数据分析结果与基于分子生物学的功能研究结果高度契合,相互支持;TNL基因与抗病信号传导基因之间频繁的“共丢失”演化模式,还指导该研究发现了多个可能参与NLR类抗病基因信号传导的新基因,开拓了从演化生物学角度挖掘抗病基因的新路径。上述发现对于深入理解植物抗病基因的演化和功能机制具有重要意义,受到评审人的一致认可,认为该研究是NLR抗病基因研究领域里程碑式的工作(This is definitely a landmark work in NLR biology)。
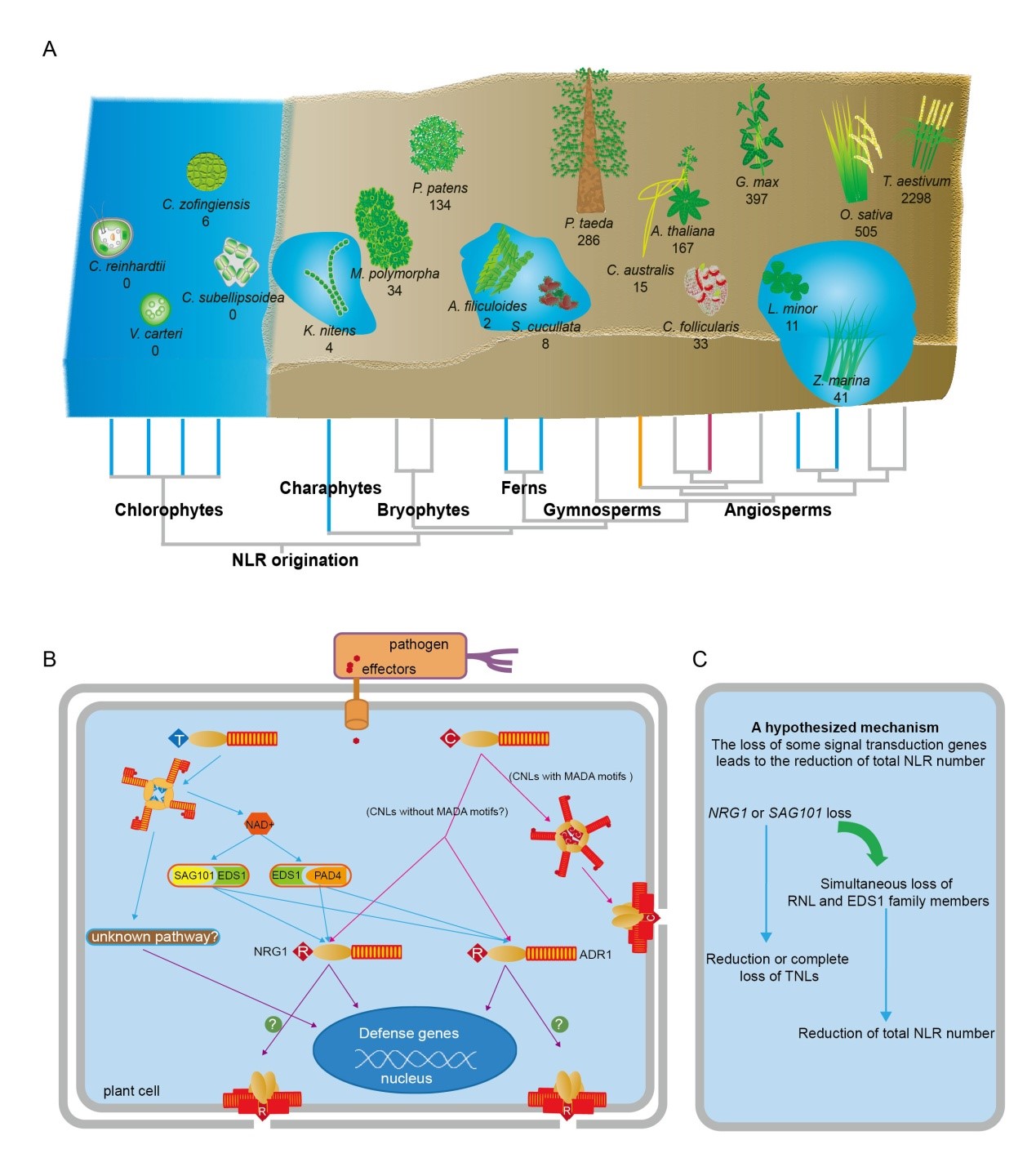
图 2 NLR基因的适应性演化模式以及分子机制示意图
该研究成果近期以 “An angiosperm NLR atlas reveals that NLR gene reduction is associated with ecological specialization and signal transduction component deletion”为题发表在Molecular Plant期刊上。37000cm威尼斯生命科学学院陈建群教授和邵珠卿副教授为该论文通讯作者,博士生刘洋为该论文的第一作者。37000cm威尼斯王强副教授、陈迪俊副教授和江苏省中国科学院植物研究所张艳梅副研究员参与了研究工作。该研究得到了国家自然科学基金和中央高校基本科研业务费的资助。
文章链接:https://www.sciencedirect.com/science/article/pii/S1674205221003142