物理学院王炜教授课题组在细胞肿瘤抑制机制的动力学及其调控方面取得重要成果,揭示出多条信号通路如何动态协作,确保细胞对促癌信号的监测和恰当响应。他们的论文“Modeling the response of a tumor-suppressive network to mitogenic and oncogenic signals”于2017年5月8日在美国科学院院报PNAS在线发表(http://dx.doi.org/10.1073/pnas.1702412114)。该工作是王炜课题组与美国Rice大学J.N. Onuchic教授等同行的合作成果,研究生田欣瑜、黄博为论文共同第一作者,刘锋教授、Onuchic教授和王炜教授为共同通讯作者。
结合基因表达调控和信号转导来研究细胞的应激反应既是细胞生物学的前沿课题,也是多学科交叉的热点课题。限于当前的实验技术条件,从单细胞水平阐明这些反应的分子机制仍然是巨大的挑战。即使能做到单细胞水平的测量,往往也只能给出某些时间点上的测量数据,而生物反应过程本身却是动态变化的。因此,建立相关生物过程的动态分子网络模型,模拟计算并刻画其动力学,从而揭示其调控机制,是重要的创新研究方式。采用这样的定量计算系统生物学研究方法,王炜课题组在细胞如何应答DNA损伤、低氧、端粒缩短等方面已经做出了系列创新工作(如PNAS,106,12245 (2009);PNAS, 108, 8990 (2011)等文章)。最近,他们提出新的模型,在细胞肿瘤抑制的动力学和调控机制方面取得重要进展。
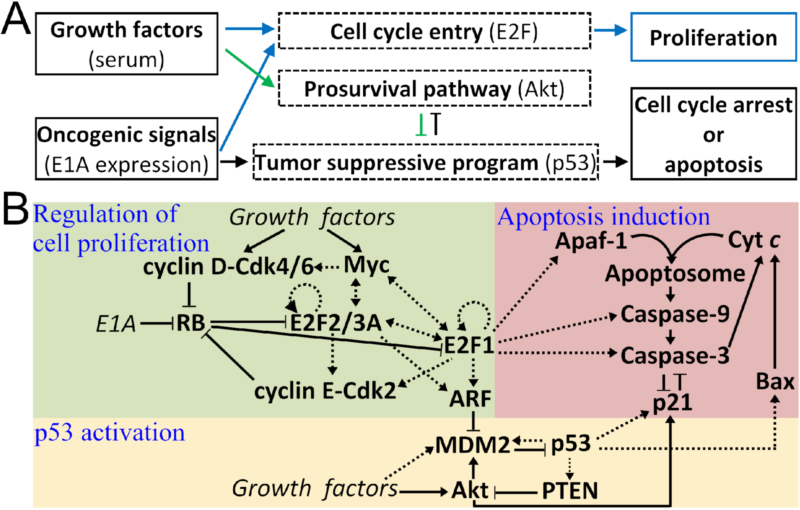
图1:细胞肿瘤抑制网络图。A)网络中的信号流;B)含3个模块的网络构成。
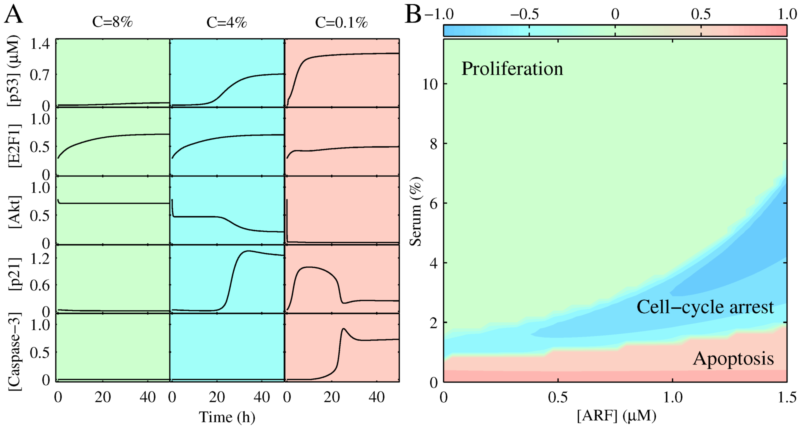
图2:信号分子动力学和细胞命运抉择。A)关键信号分子在不同血清浓度下的动力学; B)3种细胞命运的分布相图。
正常细胞含有一套精准的肿瘤抑制机制,能够识别癌信号,并通过诱导细胞周期阻断或凋亡来抑制细胞的异常增殖。尽管人们对肿瘤抑制网络的信号通路有了较明确的认识,但对其运作机制还了解甚少。其中一个基本的问题是,在异常促增殖信号(如源于E1A癌蛋白过表达)和生长因子(促细胞增殖)共同作用下,细胞是如何应答的?为此,王炜他们构建了含有三个模块的信号转导分子网络模型,分别描述细胞周期进程调控、p53活化和细胞凋亡等过程(如图1所示),开展数值模拟,刻画了网络对生长因子和E1A癌蛋白的响应特性。他们结合刻画网络的动力学,得到了一系列重要结果:1)发现网络通过ARF蛋白的“全或无”表达来区分正常和异常的促增殖信号;2)揭示了网络中重要信号分子,如p53、E2F1、Akt、p21、caspase-3等在不同情形下的动力学和功能(图2A);3)阐明了细胞命运抉择的分子机制,揭示了细胞增殖、细胞周期阻断、凋亡等不同命运对促存活和促凋亡信号强度的依赖关系(图2B);4)发现了细胞凋亡的诱导经历可逆转到不可逆转的过程,并结合细胞色素c释放和caspase-3活化的两相动力学给出了解释。这些结果给出了细胞肿瘤抑制过程的动态信号处理图像,以及蛋白质动力学对网络抑癌功能的调控机制。该工作对人们了解细胞的肿瘤抑制机制具有重要的科学意义,为恢复和调控肿瘤细胞的肿瘤抑制功能的应用研究提供了重要线索。
该工作得到国家自然科学基金、科技部973计划等项目和人工微结构科学与技术协同创新中心的支持。
(物理学院 科学技术处)