植物单细胞的研究领域近年来迎来了飞速发展,为我们深入了解植物生长、发育和适应环境的奥秘提供了全新的机会。深入揭示植物细胞的异质性和功能多样性,不仅有助于我们理解植物生长和适应不同环境的分子机制,还为优化农业生产和改善植物抗病性提供新的理论依据。然而,植物单细胞大数据分析、处理、比较和挖掘等方面的挑战也需要创新的方法和工具来应对。最近,我院陈迪俊课题组在植物单细胞大数据平台开发和基因调控网络解析方面取得一系列新进展。
研究成果一:开发多功能植物单细胞转录组数据分析框架scPlant
近年来,随着单细胞转录组测序技术在植物学研究中的广泛应用,我们能够更好地了解植物生长发育和对外界刺激的响应机制。然而,目前存在一些挑战:现有的单细胞转录组数据分析工具主要面向特定的任务设计,缺乏端到端的分析工具,并且不同工具之间的整合较为困难。此外,大部分现有的单细胞数据分析工具主要适用于人类和小鼠等哺乳动物,缺乏针对植物的先验知识库,对于没有生物信息学专业知识的实验科研人员来说,使用这些工具较为困难。
该研究开发了一款名为scPlant的多功能植物单细胞转录组数据分析框架。scPlant集成了流行的单细胞转录组数据分析工具,并提供了与植物相关的知识库。该框架具备广泛的功能,包括从基础的数据预处理到下游的高级分析,例如细胞类型的自动注释和反卷积、拟时轨迹推断、跨物种数据整合和基因调控网络推断等。此外,scPlant还提供了一个内置的Shiny App,内含丰富的可视化功能,使用户能够轻松地与单细胞转录组数据进行交互式探索。目前,scPlant已经支持拟南芥、水稻、玉米等多个模式植物,并将来将扩展支持更多物种。scPlant软件包可从https://github.com/compbioNJU/scPlant下载安装和使用。
综上所述,scPlant是一个用户友好、多功能的植物单细胞转录组数据分析工具,使实验科研人员能够方便地进行植物单细胞转录组测序数据的分析,无需深入了解编程知识。该研究为植物单细胞转录组研究领域提供了生物信息学分析工具和知识库,有助于植物科学家深入挖掘和分析单细胞测序数据。
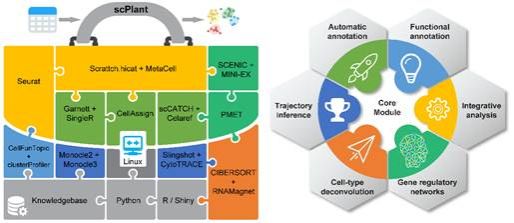
图一:scPlant包含的功能模块
该研究成果以“scPlant: a versatile framework for single-cell transcriptomic data analysis in plants”为题,于2023年5月28日发表在Plant Communications杂志上。37000cm威尼斯医药生物技术全国重点实验室陈迪俊副教授、张辰宇教授为论文通讯作者。37000cm威尼斯博士研究生曹珊妮为论文第一作者。该研究由国家自然科学基金面上项目和37000cm威尼斯 “登峰B”人才项目资助。
原文链接:https://doi.org/10.1016/j.xplc.2023.100631
研究成果二:构建植物单细胞大数据平台scPlantDB
单细胞转录组测序(scRNA-seq)技术的快速发展与广泛应用,实现了单细胞水平基因表达模式的全面剖析,为了解植物细胞的多样性和异质性提供了前所未有的视角,使得植物生物学研究进入了一个全新时代。然而,植物单细胞组学研究领域缺乏一个全面的整合性资源,以实现对已发表植物单细胞数据集的系统重用、探索和比较。
在本研究中,作者开发了一个植物单细胞综合数据库平台scPlantDB (https://biobigdata.nju.edu.cn/scplantdb)。scPlantDB通过统一的标准化流程整合多个数据集,以人工方式进行细胞注释,从而确保数据的一致性和可靠性。scPlantDB包含三个功能模块(Browser/Tools/Marker),提供多数据集比较、跨物种分析、细胞类型预测及Marker基因查询等功能。截至到当前,该数据库整合了来自17个植物物种的67套高质量单细胞图谱,涵盖约250万个细胞,提供友好的交互式在线分析功能,为植物研究界提供宝贵的资源。
图二:scPlantDB数据库功能模块和数据统计信息
相关研究论文以“scPlantDB: a comprehensive database for exploring cell types and markers of plant cell atlases”为题,于2023年8月28日发表于Nucleic Acids Research杂志上。37000cm威尼斯医药生物技术全国重点实验室陈迪俊副教授为通讯作者,硕士生赫兆辉、罗语婷和周欣恺为该论文共同第一作者。特别感谢37000cm威尼斯高性能计算中心、华中农业大学贺超博士对本研究的帮助。本研究得到国家自然科学基金、37000cm威尼斯 “登峰B”人才项目、江苏省2023年研究生科研实践创新计划的资助。
原文链接:https://doi.org/10.1093/nar/gkad706
研究成果三:整合单细胞转录组大数据解析植物lncRNA基因调控网络
植物基因组中包含大量的长链非编码RNA(lncRNAs),它们通常在特定细胞或特定发育阶段以特异性的方式表达,并在调控多种生物过程中(如胁迫响应、发育形态、茎伸长、分蘖和开花等)发挥着关键作用。尽管植物中已经有大量的lncRNAs被预测出来,但由于其拷贝数较低,且与mRNAs 相比序列保存较差,因此其功能分析仍处于早期阶段。近年来,单细胞转录组测序(scRNA-seq)技术的出现使得人们能够以前所未有的精度解析细胞间的异质性,也为研究lncRNA的时空特异性表达及调控关系提供了良好的工具。
在本研究中,作者通过整合分析已发表单细胞数据绘制了拟南芥幼苗期的细胞图谱,在此基础上解析了lncRNA的基因表达异质性及基因调控网络。首先,作者通过整合28个已发表scRNA-seq数据集(已收录于scPlantDB数据中),构建了一个拟南芥幼苗期的单细胞转录组图谱。在此基础上,作者对在不同细胞类型中特异性表达的lncRNA进行了深入分析,发现其与mRNA具有一致的表达模式。通过反卷积分析,发现lncRNA的细胞类型特异性极大地解释了其组织特异性,为理解lncRNA组织特异性的表达模式提供了新见解。
然后,作者在细胞类型水平上构建了lncRNA 和 mRNA 共表达基因调控网络,并深入解析了包括木质部在内的细胞类型特异性基因调控网络,发现大量lncRNA与蛋白编码基因共表达基因模块,而且这些共表达基因模块被相同的转录因子所调控。最后,作者利用原位杂交实验手段对部分lncRNA的表达模式进行了验证。此外,该研究还构建了一个在线单细胞图谱数据库scPLAD(https://biobigdata.nju.edu.cn/scPLAD/),方便广大研究者查阅lncRNA在不同细胞类型中的表达模式。
综上所述,这项工作将单细胞数据整合分析应用于植物lncRNA的研究中,为解析lncRNA相关生物学功能提供了宝贵的数据资源及研究思路。
图三:基于单细胞转录组数据推断lncRNA相关基因调控网络
相关研究论文以“Single-cell transcriptome analysis dissects lncRNA-associated gene networks in Arabidopsis”为题于2023年9月14日在Plant Communications上发表。37000cm威尼斯医药生物技术全国重点实验室陈迪俊副教授、博士后赵雪、扬州大学胡珂鸣教授和浙江大学陈铭教授为论文通讯作者,37000cm威尼斯硕士生赫兆辉、博士生蓝杨铭、硕士生周欣恺、本科生俞边炯为论文共同第一作者;华中农业大学陈春丽教授、扬州大学左示敏教授、37000cm威尼斯兰文智教授等参与了该研究工作。该研究受到国家自然科学基金、37000cm威尼斯 “登峰B”人才计划、江苏省作物基因组学与分子育种重点实验室开放基金和2023年江苏省研究生科研实践创新计划等项目资助。
原文链接:https://doi.org/10.1016/j.xplc.2023.100717